※学習初学者の勉強のアウトプットですので、もしコードや解釈に間違い等あればご指摘頂けますと幸いです。
メモも兼ねて、各コードなるべく詳しく説明をつけるようにしています。
Contents
ハイパーパラメーターの調整について
機械学習においてハイパーパラメーターの調整はとても重要ですが、ハイパーパラメーターの種類はたくさんあり、これを手動で毎回変更するのは大変です。
ハイパーパラメーターの範囲を指定して、精度の良いハイパーパラメーターを自動で見つけてもらう方法があります。主な方法としては、グリッドサーチとランダムサーチの2種類があります。
グリッドサーチ:
調整したいハイパーパラメーターの値の候補を明示的に複数指定し、ハイパーパラメーターセットを作成します。モデルの評価を繰り返すことにより、最適なハイパーパラメーターセットが作成されます。値の候補を明示的に指定するため、ハイパーパラメーターの値に文字列や整数、True/Falseといった数学的に連続ではない値をとるハイパーパラメータの探索に向いている一方、多数のハイパーパラメーターをチューニングするのは不向きです。
ランダムサーチ:
ランダムサーチは、ハイパーパラメーターが取りうる値の範囲を指定し、確率で決定されたハイパーパラメーターセットを用いてモデルの評価を行うことを繰り返し、最適なパラメーターを探す手法です。値の範囲は、ハイパーパラメーターの確率関数を指定します。
グリッドサーチ
scikit-learn内の乳がんのデータセットを使用し、グリッドサーチにてハイパーパラメーターの調整を行ってみます。
#必要なライブラリのインポート
import scipy.stats
from sklearn.datasets import load_breast_cancer
from sklearn.svm import SVC
from sklearn.model_selection import GridSearchCV
from sklearn.model_selection import train_test_split
from sklearn.metrics import accuracy_score
#乳がんのデータセットを取得し、中身を確認する
cancer = load_breast_cancer()
#X,yにそれぞれ説明変数と目的変数を格納する
X = cancer.data
#yにtargetを格納する
y = cancer.target
#train_data ,testデータに分割する
train_X, test_X, train_y, test_y = train_test_split(X, y, test_size = 0.3, random_state=42)
#ハイパーパラメータの値の候補を設定する
model_param_set_grid = {SVC(): {"kernel": ["linear", "poly", "rbf", " sigmoid"],
"C": [10** i for i in range (-5,5)],
"decision_function_shape": ["ovr", "ovo"],
"random_state":[42]}}
max_score = 0
best_param = None
#グリッドサーチでハイパーパラメーターを検索する
for model, param in model_param_set_grid.items():
clf =GridSearchCV(model, param)
clf.fit(train_X, train_y)
pred_y = clf.predict(test_X)
score = accuracy_score(test_y, pred_y)
if max_score < score:
max_score = score
best_param = clf.best_params_
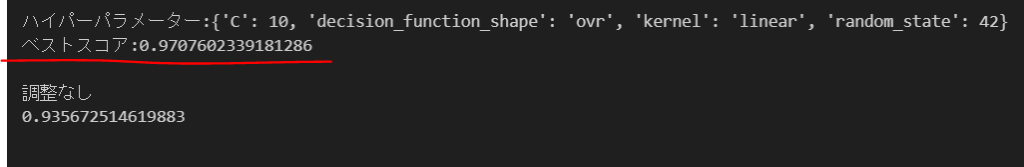
print(f"ハイパーパラメーター:{best_param}")
print(f"ベストスコア:{max_score}")
svm = SVC()
svm.fit(train_X, train_y)
print()
print('調整なし')
print(svm.score(test_X, test_y))解説
#必要なライブラリのインポート
import scipy.stats
from sklearn.datasets import load_breast_cancer
from sklearn.svm import SVC
from sklearn.model_selection import GridSearchCV
from sklearn.model_selection import train_test_split
from sklearn.metrics import accuracy_score
#乳がんのデータセットを取得し、中身を確認する
cancer = load_breast_cancer()
#X,yにそれぞれ説明変数と目的変数を格納する
X = cancer.data
#yにtargetを格納する
y = cancer.target
#train_data ,testデータに分割する
train_X, test_X, train_y, test_y = train_test_split(X, y, test_size = 0.3, random_state=42)まず必要なライブラリをインポートし、X,yにそれぞれ説明変数、目的変数を格納し、訓練データとテストデータに分けます。
#ハイパーパラメータの値の候補を設定する
model_param_set_grid = {SVC(): {"kernel": ["linear", "poly", "rbf", " sigmoid"],
"C": [10** i for i in range (-5,5)],
"decision_function_shape": ["ovr", "ovo"],
"random_state":[42]}}
max_score = 0
best_param = None今回はサポートベクターマシンでのパラメーター最適化を行ってみます。
まず、"kernel"ではSVCモデルのカーネル関数の種類を指定しています。
lenear: 線形カーネル、線形の決定境界を生成します。
poly: 多項式カーネル、非線形な決定境界を生成します。
rbf : 動径基底関数(RBF)カーネル、無次元の特徴空間に写像し、非線形な決定境界を生成します。
sigmoid: シグモイドカーネル、シグモイド関数を用いて非線形空間に写像します。通常、ニューラルネットワークの活性化関数に似た形状を持つ決定境界を生成します。
"C"は正則化パラメータを調整します。SVMの過学習を防ぐためのパラメータです。
"decision_function_shape"は、分類手法を指定しています。SVMは通常二値分類ですが、複数クラスある場合は多クラス分類に拡張する櫃よぅがあります。"ovr"(One vs Rest)は一つのクラスとそれ以外という分類を行い、"ovo"(One vs One)はクラスを2つで1つの組み合わせにし、その全ての場合に対して各データを分類する方法です。
"random_state"を設定することにより、再現性を確保できます。任意の整数値を指定します。
#グリッドサーチでハイパーパラメーターを検索する
for model, param in model_param_set_grid.items():
clf =GridSearchCV(model, param)
clf.fit(train_X, train_y)
pred_y = clf.predict(test_X)
score = accuracy_score(test_y, pred_y)
if max_score < score:
max_score = score
best_param = clf.best_params_model_param_set_gridにはモデル名とそのハイパーパラメータの候補が設定されていますが、.items()でモデル名をmodelに、パラメーターの候補をparamに格納します。それらをGridSearchCVに入れてグリッドサーチを実行し、最適なハイパーパラメーターを探索していきます。
各モデルに対してグリッドサーチを行った後、テストデータを使用して予測を行いスコアを出し、最も精度の高いモデルとパラメーターの組み合わせをbest_paramに格納します。
print(f"ハイパーパラメーター:{best_param}")
print(f"ベストスコア:{max_score}")
svm = SVC()
svm.fit(train_X, train_y)
print()
print('調整なし')
print(svm.score(test_X, test_y))グリッドサーチで探索した最適値と、その最適値を用いたモデルのスコアを表示してみます。
また、ハイパーパラメーターを何も調整しなかった場合のスコアも出してみます。
グリッドサーチで探索したハイパーパラメーターを使用した方が、スコアが高いことがわかります。